论文阅读:单细胞基因调控网络 SCRN
基本信息
title: SCRN: Single-Cell Gene Regulatory Network Identification in Alzheimer’s Disease
date: NOVEMBER/DECEMBER2024
前置知识
这篇论文使用了DGCNN作为主要的神经网络
DGCNN & sortpooling:https://blog.csdn.net/yyl424525/article/details/112340706
注意这个DGCNN全称是DeepGraphConvolutional Neural Network,如下的这个并不是本文使用的:
DGCNN(Dynamic Graph CNN for Learning on Point Clouds)
虽然两个名字一样,并且都是作用于图结构的神经网络,并且结构还有一定的相似性,但这两个不是一个东西!
背景
阿尔兹海默疾病与基因有很大关系,但完整的基因调控网络非常复杂,难以实验得到。因此,这篇论文尝试利用单细胞数据来构建完整的阿尔兹海默疾病相关的基因调控网络。数据集是 基因的表达量、已知的基因调控关系。我们希望使用这部分数据来推断完整的基因调控网络关系。
困难:
- 基因调控网络非常庞大,难以全部计算。
- 不同基因的局部网络节点数不同。
- 阿尔兹海默患者与正常人的GRN不同。
主要结构
这个神经网络做的工作是link prediction,判断这两个基因是否存在调控关系。
其实这个结构主要是借鉴这篇论文 Link Prediction Based on Graph Neural Networks:https://arxiv.org/abs/1802.09691

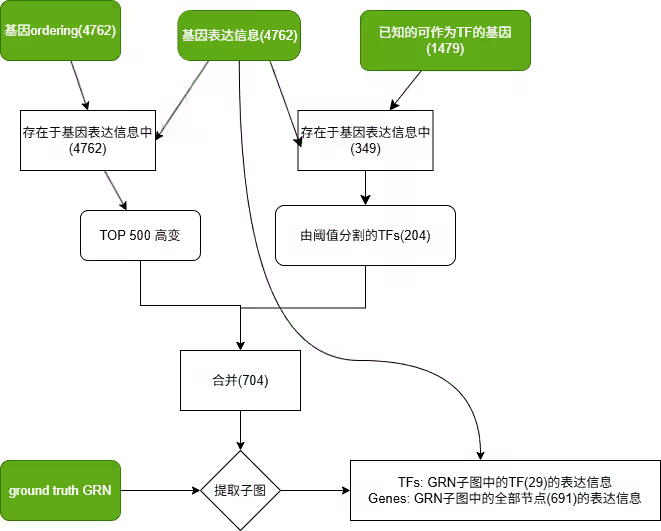
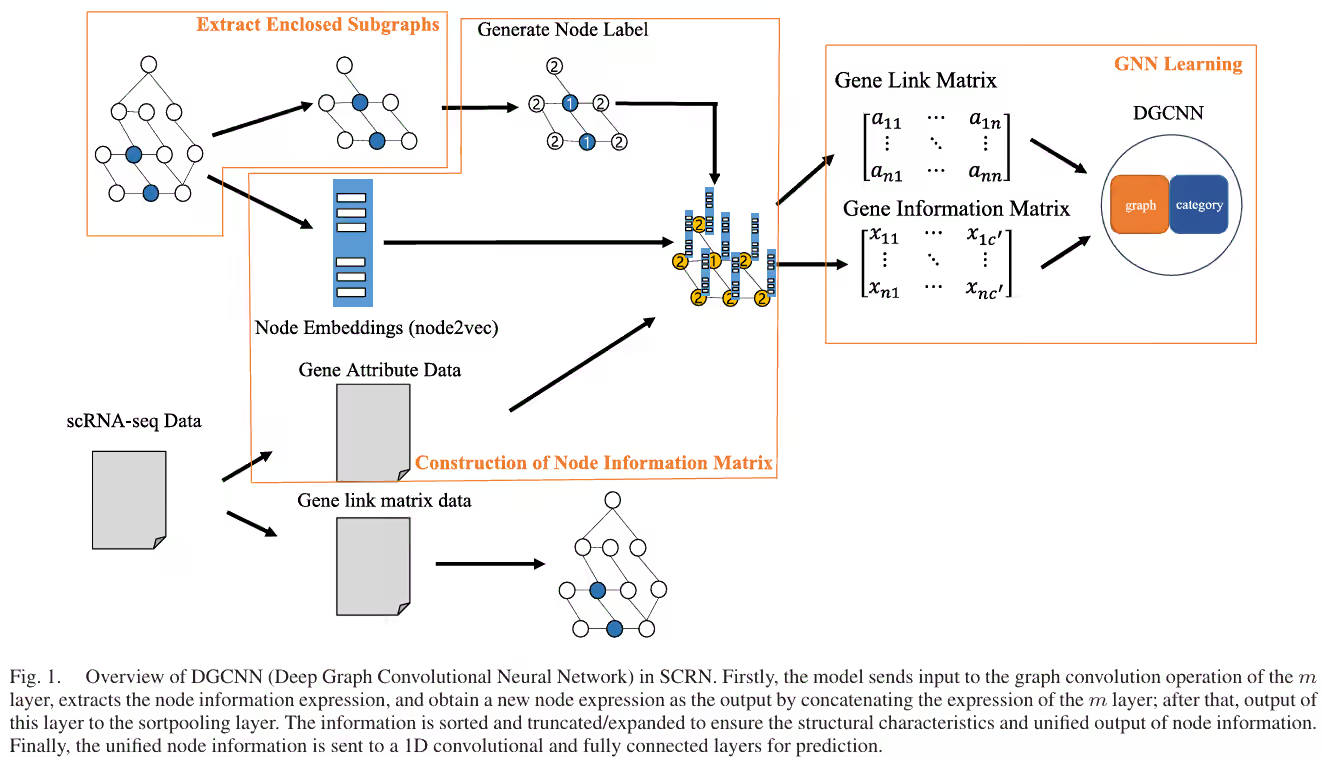
构建子图
首先先给出我们需要判断的两个基因。
由于整个基因调控网络非常庞大,难以全部计算。并且根据局部性原理,我们仅需要考虑与中心基因k跳距离内的基因。
将这k跳距离内的基因构建一个封闭子图。
构建DGCNN的输入
DGCNN的输入包括2个部分:
- 基因邻接矩阵(gene link matrix)
- 基因信息矩阵(gene infomation matrix)
note:为了更好地让模型知道中心基因是哪一个,需要将中心基因的位置固定在 基因信息矩阵 中。这篇论文将中心基因的位置固定为前两个元素。
而基因信息矩阵又由3个部分拼接而成:
- node label (结构重要性)
- node attribute (基因表达信息)
- node embedding (拓扑信息)
node label
这个值是一个标量,文中使用双半径节点标记方法计算。(来自论文 Link Prediction Based on Graph Neural Networks)
提出的背景是:如果不做特殊标记,当GNN处理一个子图时,它难以区分哪个节点是我们要预测链接的“中心节点”(即节点x和y)。它会将所有节点一视同仁,从而丢失重要的结构信息。
主要思路是:标签值越小,表示该节点在结构上越重要(离中心节点越近)
node attribute
这也是一个标量,代表这个基因的表达量。
node embedding
这个embedding是一个向量,仅包含图结构的拓扑信息,通过node2vec计算得来。
DGCNN
这里简要过一下DGCNN。DGCNN可以将离散的、数量不定的图结构转换为连续的、固定长度的稠密向量表示,从而可以CNN处理。

这里再贴一张DGCNN原论文的图:

每一个图卷积能将节点消息进行一个变换(此处论文原文存在笔误,把形状写错了):
其中,是节点特征矩阵,是权重矩阵,是加了自环的邻接矩阵,是的度矩阵。
图卷积的叠加模拟了WL颜色编码,同时利用神经网络强大的拟合能力,使得其更能代表节点的重要性。
然后通过sortpooling层,将节点的embedding截取前K个并拼接,得到一个固定大小的向量表示()。
经过一维卷积(卷积核、步长均为,即对每个节点的内部信息进行卷积),再经过一个MLP,最终输出一个的classificaion,代表有边还是没边。
训练过程
- 从已知的基因调控网络中,选择两个基因作为中心基因(移除这两个基因的边,如果有的话),构建输入
- 传入神经网络,得到一个分类输出
- 使用交叉熵损失函数进行优化。
实验结果
实验结果达到了state-of-the-art的水平。
通过这个方法,将两两基因进行计算,推断了完整的基因调控网络。发现阿尔兹海默患者(AD)的GRN与正常对照组(CT)有很大不同。