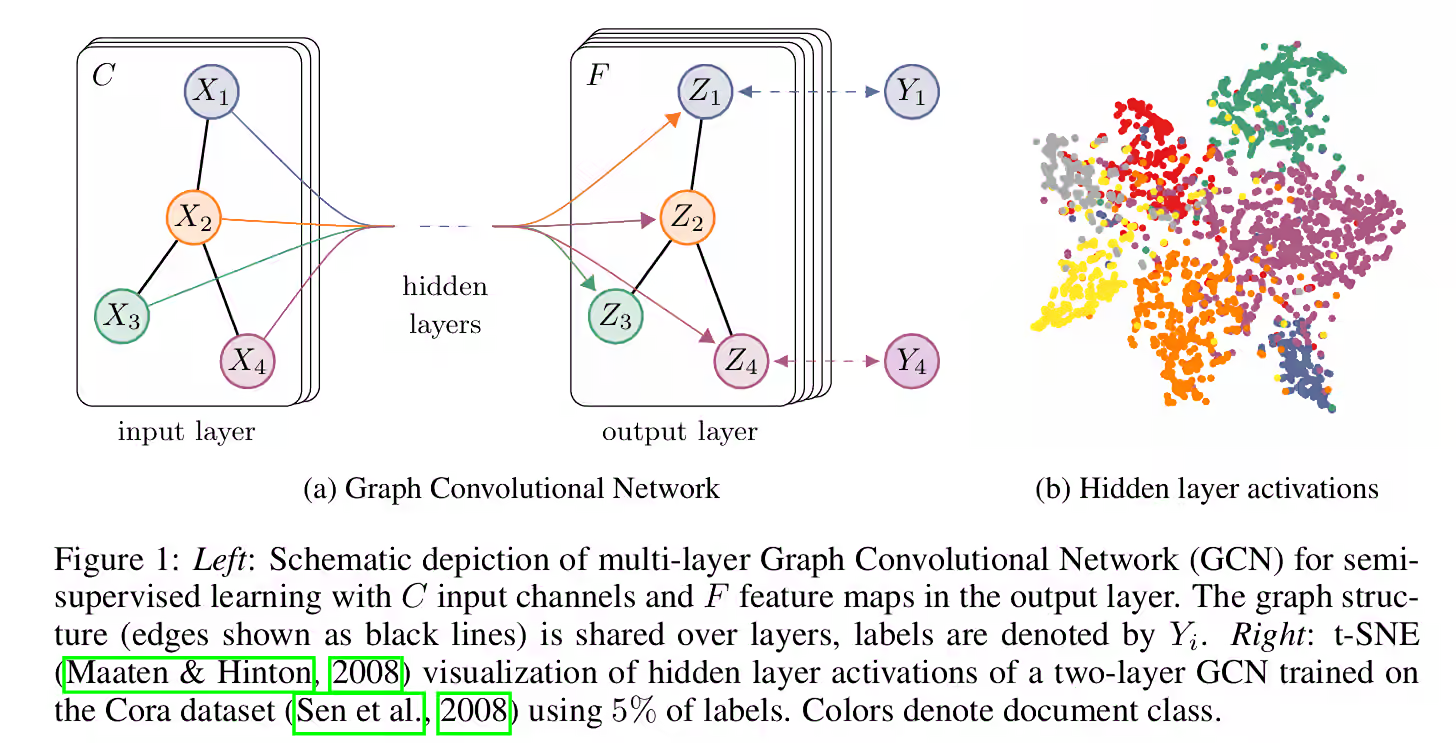
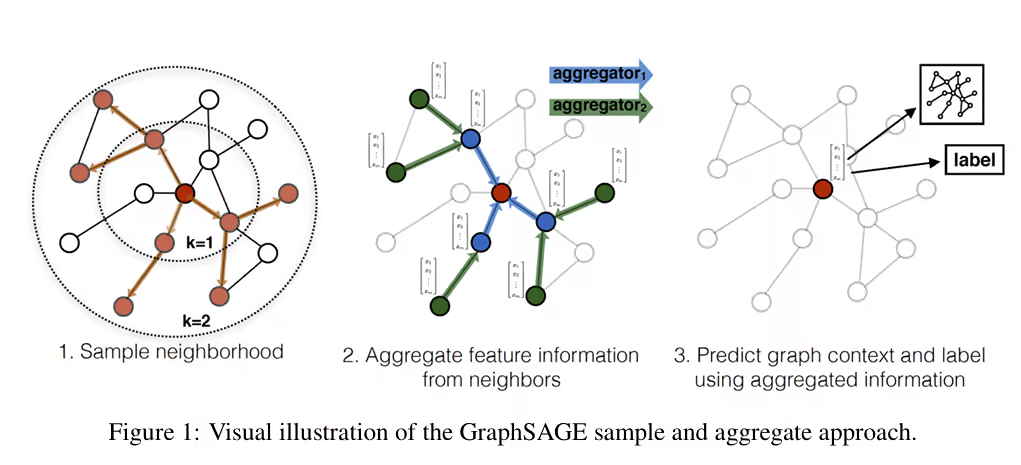
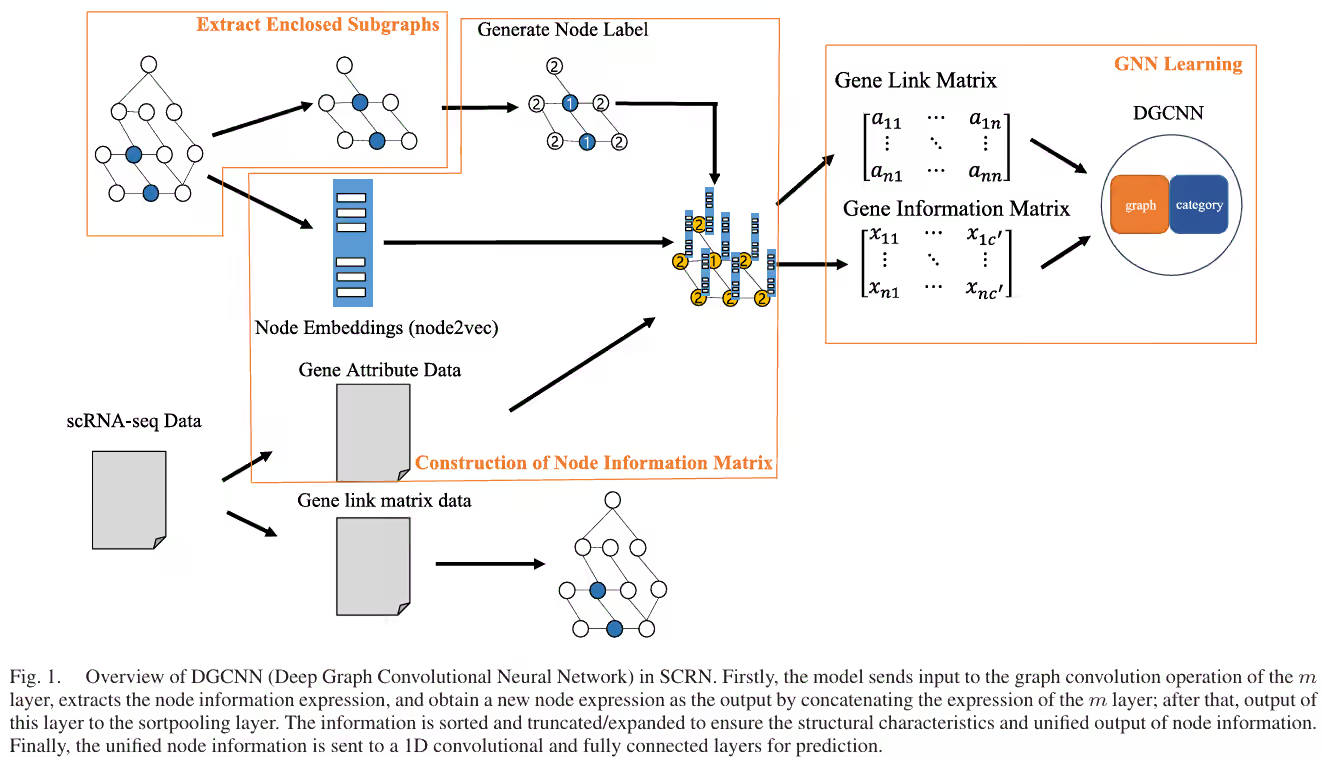
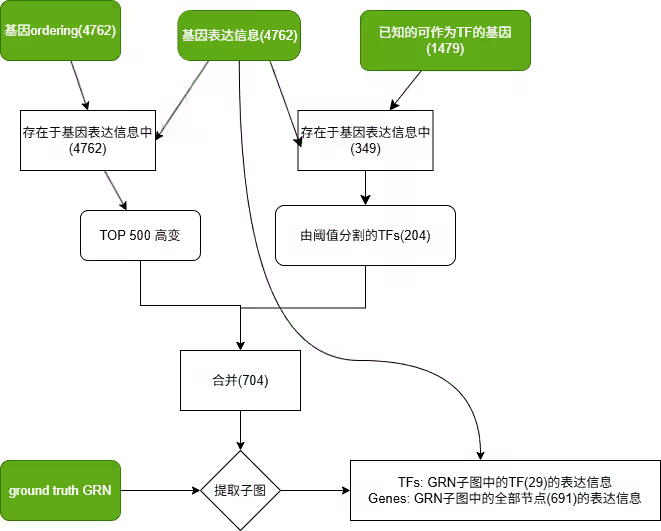
论文阅读:LineGRN 线图神经网络推断基因调控网络
basic info
title: LineGRN: a line graph neural network for gene regulatory network inference
date: not final version till now
背景
由于基因调控网络很大且已知的调控关系数量有限,这就带来了一个严重的问题:节点的度很低
为了解决节点度数低,模型难以挖掘信息的问题,LineGRN提出了一种巧妙地方法来使得图变得稠密。
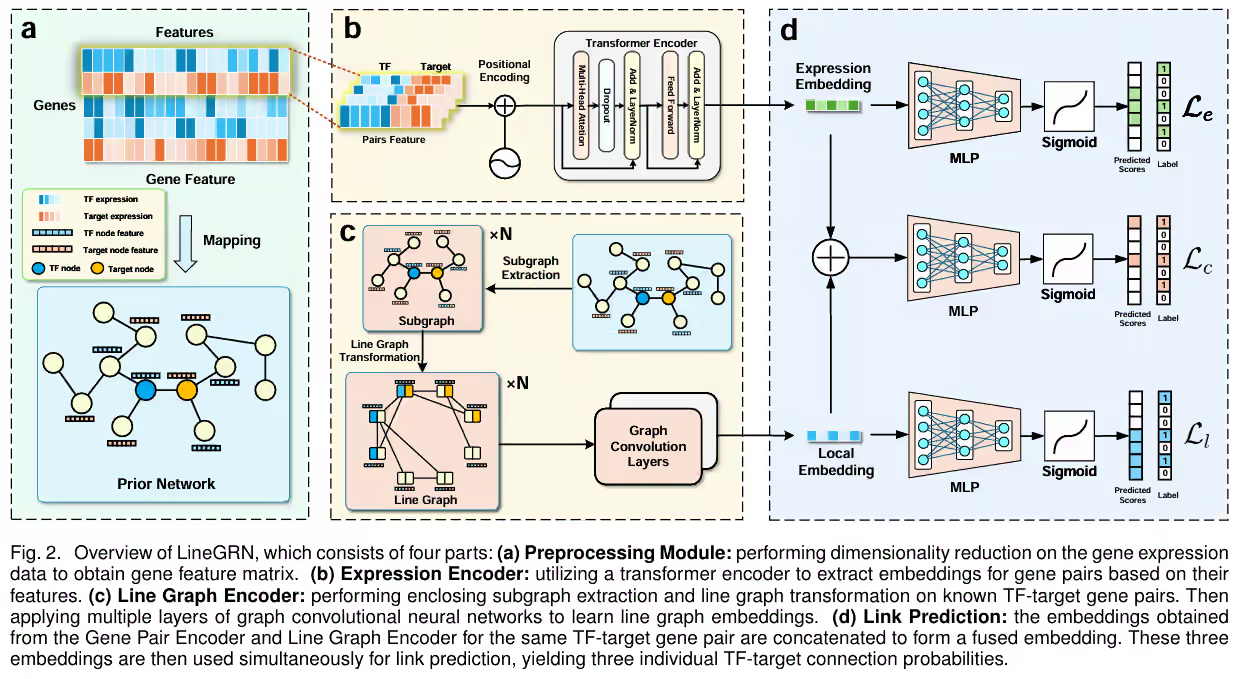
整体架构

模型主要由两部分构成:GRN特征提取、基因表达特征提取。
- 基因表达特征提取 的输入是目标基因对。
- GRN特征提取 的输入是目标基因对,以及局部GRN图。
基因表达特征
预处理
值得注意的是,由于我们想要提取的目标是基因。因此,batch对应的维度就是基因。
而由于细胞之间具有相似性,可以先利用VAE来提取基因表达的低维特征。
这样,神经网络可以学习到每个基因的表达特征,得到一个低维的基因表达特征矩阵:
特征融合
为了增强融合基因对的表达特征,LineGRN采用 这篇论文中提出的subvector方法(STGRNS: aninterpretable transformer-based method for inferring gene regulatory networks from single-celltranscriptomic data)
为了能让模型观察到局部特征之间的联系,并没有直接采用concat进行拼接,而采用了一种叫做subvector的方法。
而subvector的方法是将基因表达特征分成多个子向量,每个子向量的长度为w。然后将子向量进行拼接,最终得到整个向量,这样能让模型捕捉到局部的特征。
再对这个向量进行positional encoding,就可以送进transformer中进行编码,然后输出一个特征融合向量。
GRN特征提取
line graph
为了减少不必要的计算,LineGRN只考虑了局部GRN图中的基因对,原文中考虑了目标基因对的2-hop子图。
这篇论文提出了一个很有趣的方法来增强GRN特征提取的能力,就是将其进行 line graph 化。

line graph会将边转化为节点,节点之间的连接关系就是原图中的边。
由于最终我们需要目标基因对的表达,所以必须确保存在这个节点,因此需要认为地给这两个基因之间添加一条线。
这样,就能让原来稀疏的GRN图变得稠密,从而提高模型的表达能力。

如果新节点由合并得到,那么这个新节点的特征就是原节点的拼接,即:
GNN
这个GNN采用了比较朴实无华的方式来应对可变邻居数量,每一层GNN将周围一阶邻居的特征进行简单的融合:
通过堆叠多层GNN,就可以提取到GRN拓扑信息。
为了防止特征在融合的过程中消失,也同时为了易于训练,LineGRN使用了残差连接,将每一层的输出进行拼接:
当然,我们只关心这部分中目标基因对的表示。
loss
记GNN最终输出的目标基因对的特征为,transfomer的输出为。
那么两部分的融合记为
为了能让这两部分网络都得到良好的训练,如图中D部分所示:

这3个特征都输入到各自的MLP中,进行预测任务,然后使用二元交叉熵计算loss,得到。
由于并没有完整的信息,因此是偏高的,可以使用超参数来平衡这两个loss。
最终总loss如下:
*:原文中符号用的有点混乱,我将一些符号就行了替换
结果
lineGRN展现出了强大的鲁棒性,即便在有边扰动的情况下,远远高出了同类的其他模型