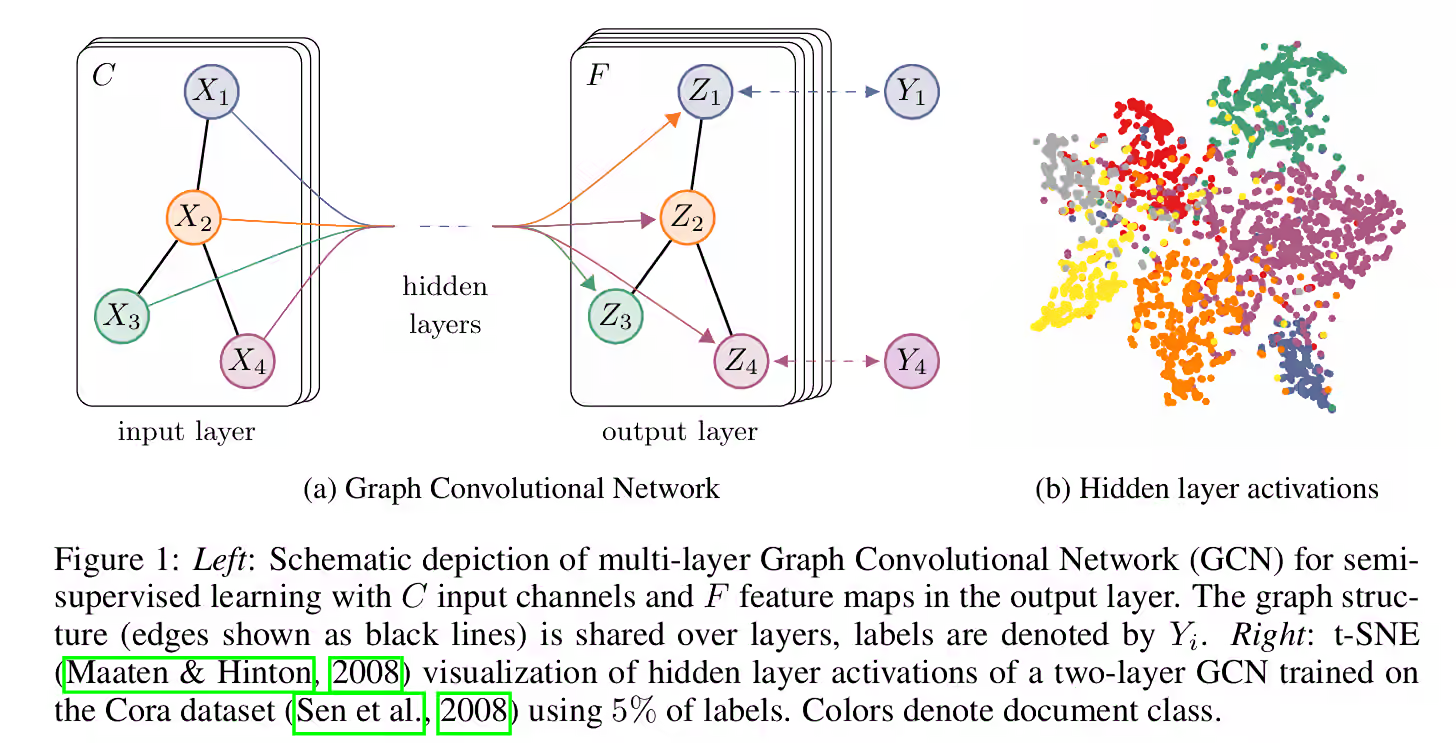
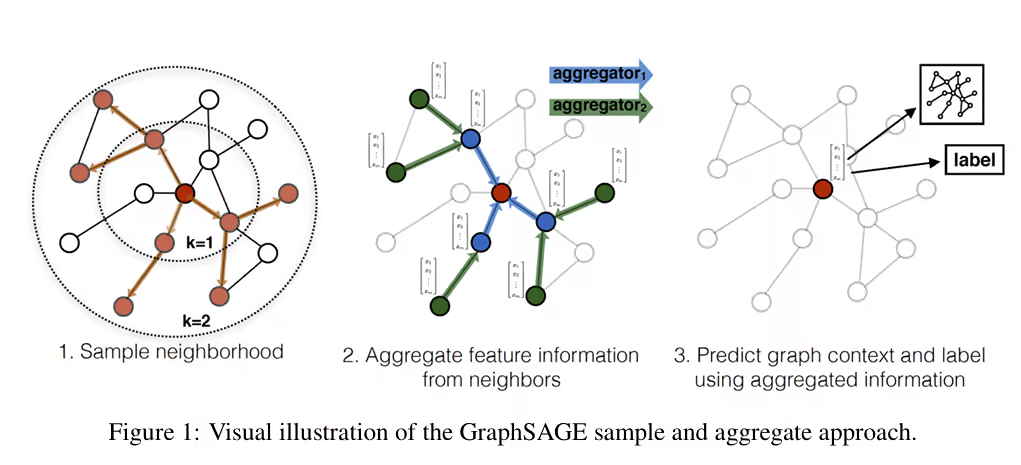
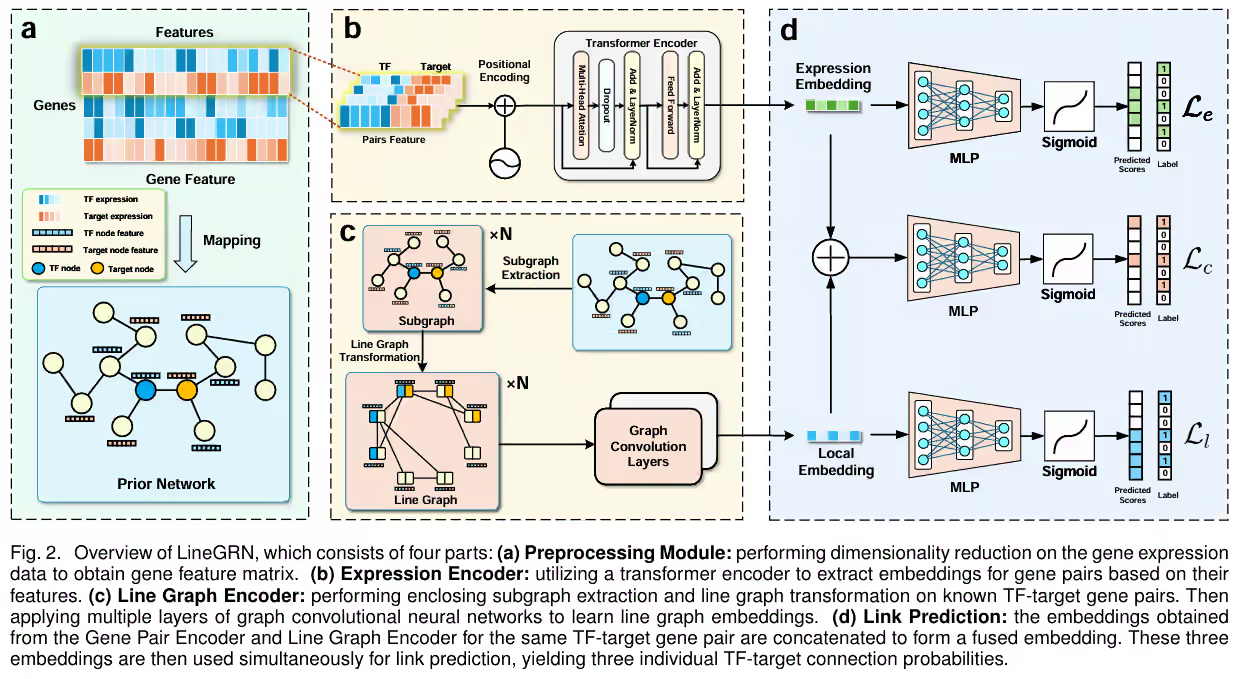
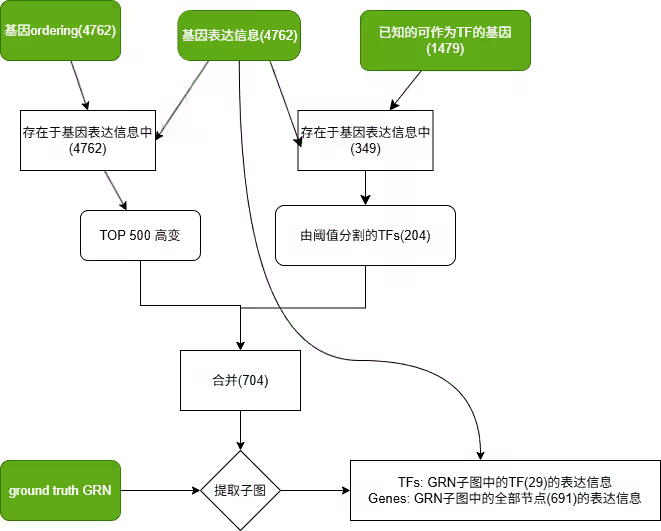
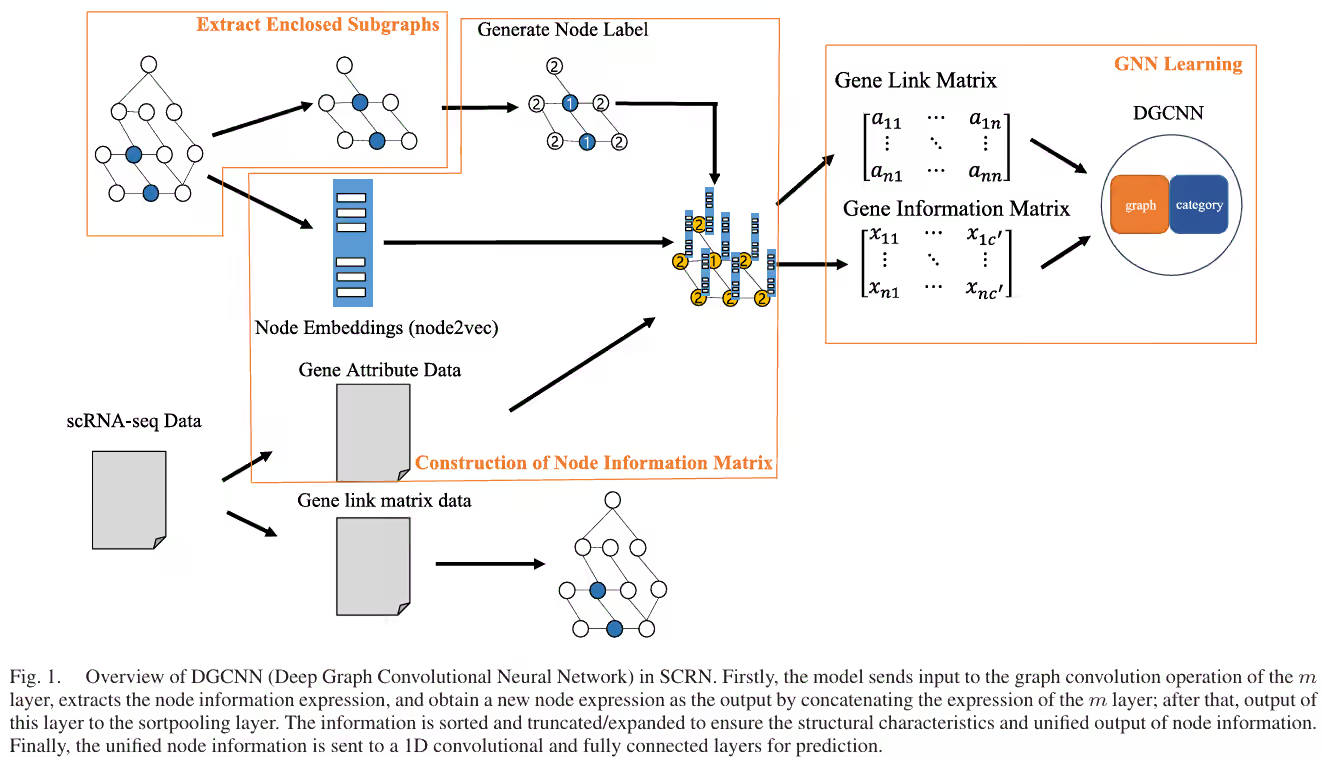
GRN多元的实验结果
数据集
数据集主要分为以下4种类型:
- Non-specific xxx-seq: 非特异性的数据。
- Specific xxx-seq: 针对某种细胞的特异性数据
- STRING:蛋白质-蛋白质相互作用数据库。可视为权威。
- LOF/GOF:基于基于敲除或基因过量表达的实验数据。可视为权威。
scATAC-seq :染色质的开放程度。
ChIP-seq:蛋白与基因关系的实验数据。
特异性
同一种基因在不同的细胞中可能具有不一样的调控关系。常常使用TFs+xx来表示top xx个特异性的TFs。
如TFs+1000表示最具有特异性的1000个TFs。
细胞类型
m开头的一般指老鼠(mouse),如:mDC、mESC
h开头的一般指人类(human),如:hESC、hDC
人造数据集
如果数据集不够多,可以使用算法生成假的GRN。
可视化技术
UMAP
这是一种在2018年提出的图降维算法,通过拓扑理论同时兼顾数据的局部和全局结构。
基因表达量

同一种基因在不同的状态下的表达量不同,可以依次作为依据认为其具有特异性。
局部GRN
直接将部分GRN画出来。关联性越强,其中的线条就越粗。
评价指标
- AUROC:区分正负样本的能力
- AUPRC:应对正负样本不平衡的能力
- MCC
- F1,recall,precision,accuracy
KL散度
对于正负样本分布的问题,可以用KL散度来评估模型的预测能力。
超参数分析
如果超参数在2维及以下,可以画出超参数-评价指标的图。
如果超参数过多,就作为一维指标去画。
消融实验
使用5折交叉验证方法进一步增强说服力。并且这部分可以结合评价指标进行图示。
将提出的方法与其他方法进行对比
将state-of-the-art的方法进行对比

其中w/o指without

画出箱形图,将置信度一起展示。
将提出的方法与去除该方法进行对比

将提出的模型进行变体,如图中的LineGRN-就表示去除线图化操作。

通过这种排列组合的方法就能画出一堆图。
鲁棒性实验/敏感度实验
- 针对不同的数据集
- 边扰动:随机添加或删除边
- 节点扰动:随机添加或删除节点
并且与已有的方法进行对比。还可以根据扰动的幅度做出图表。

可以做出维度分析图
示例展示/案例研究
选择一个具体的示例,将模型的输出与权威的结果进行对比。
eg. 将计算出来的调控关系与已知的调控关系进行对比
应用实际:生物学应用
对尚未解决的问题,给出模型的输出结果
- 靶向药品的预测
- 基因疾病的分析(不同的表达量)
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来源 57U's Blog!
评论